*Uniwersytet Cambridge
W październiku ubiegłego roku na okładce magazynu Nature pojawiła się mucha. Nie dla wszystkich może to być oczywiste, bo widzimy tam coś, co wygląda jak plątanina niezliczonej ilości kolorowych kabli; na czarnym tle przypomina raczej wizualizację zderzenia ciał niebieskich, a nie organ tego niepozornego owada. Tytuł ogłaszał zakończenie ambitnego projektu, który pozwolił skonturować kompletną mapę mózgu muszki owocówki z gatunku Drosophila melanogaster. Dlaczego owad, którego z utrapieniem odganiamy od naszych jabłek i bananów, zasłużył na wyróżnienie w prestiżowym magazynie naukowym?
Wyjaśnienie rozpoczniemy od banalnego stwierdzenia, że aby nie zgubić się w obcym mieście, dobrze jest mieć mapę. Aby trafić pod konkretny adres, najlepiej, jeśli mapa ta jest dobrze oznaczona. Analogicznie neuronaukowcy, chcąc zrozumieć szczegółowe działanie mózgu, potrzebują dokładnej mapy jego struktur. Typowy neuron w ludzkim mózgu ma około 12 mikrometrów średnicy, a komórek tych jest około 86 miliardów, sprawa zatem nie jest prosta. Tym bardziej, że do tej pory jedyna kompletna mapa układu nerwowego organizmu żywego pochodziła z małego robaczka C. elegans, który ma dokładnie 302 neurony. Przeskok ilościowy wydaje się zatem ogromny.
Naukowcy zainteresowali się więc wspomnianą Drosophilą. Chociaż muszek owocówek nie lubimy w naszej kuchni, okazują się one bardzo przydatne w laboratorium biologicznym, szczególnie w badaniach genetycznych. Ich mózg liczy około 140 000 neuronów, co czyni problem istotnie trudnym, lecz nie niemożliwym do zaatakowania przy użyciu współczesnej technologii. Zrzeszeni w konsorcjum badacze z ośrodków Cambridge, Harvard, Princeton i Centrum Badań Janelia zakasali rękawy i rozpoczęli intensywne prace w celu stworzenia, przy współpracy z kilkoma firmami, szczegółowej mapy neuronów oraz ich połączeń; tak zwanego konektomu.
Współczesna biologia pełna jest -omów (czyli szczegółowych baz danych): genomów, proteomów, regulomów etc.
Wróćmy do metafory mapy. Aby stworzyć od zera mapę geograficzną, warto zacząć od topografii terenu, na przykład zdjęć satelitarnych. Zamiast satelity do obrazowania neuronów będziemy potrzebować mikroskopu elektronowego, dzięki któremu możemy uzyskać obrazy o wysokiej rozdzielczości. Działo elektronowe wytwarza strumień ujemnie naładowanych cząstek, które są przyspieszane i skupiane w wąską wiązkę. Dodatkowe soczewki magnetyczne skupiają wiązkę elektronów na próbce tkanki biologicznej. Na podstawie stopnia penetracji elektronów przez tkankę można stworzyć obraz organelli komórkowych. Tkankę trzeba wcześniej odpowiednio przygotować: często przy użyciu laserów, które precyzyjnie tną ją na ultracienkie fragmenty (o grubości około 60 nanometrów). Po przeskanowaniu rozłącznych fragmentów tkanek konieczna jest ich ponowna integracja, czyli połączenie w trójwymiarową strukturę (czasem wymaga to dodatkowego przekształcania obrazów, np. skalowania czy ściskania).
W kolejnym kroku na zdjęciach satelitarnych warto zaznaczyć poszczególne obiekty, takie jak ulice, rzeki, budynki, tereny zielone. W przypadku konektomu jesteśmy głównie zainteresowani kształtem neuronów oraz połączeniami między nimi. Do efektywnej analizy olbrzymiego zbioru danych niezbędne są narzędzia sztucznej inteligencji, które pomagają w szybkiej segmentacji struktur neuronowych z obrazów mikroskopowych poprzez identyfikację granic tkanek i rekonstrukcję trójwymiarowych morfologii, tzw. ,,szkieletów”. W szczególności pomocny w tym procesie jest algorytm wypełniania konturowego (ang. flood-filling) wspomagany przez konwolucyjne sieci neuronowe. Ta unikalna architektura umożliwia sieciom neuronowym iteracyjne rozszerzanie poszczególnych fragmentów tkanek biologicznych w jednolite trójwymiarowe obiekty. Inne algorytmy, również oparte na sieciach konwolucyjnych, pozwalają w szybki i dokładny sposób wykryć synapsy, czyli połączenia pomiędzy neuronami (biologicznymi), oraz związane z nimi neuroprzekaźniki (chemiczne transmitery kontrolujące przesyłanie informacji).
Rekonstrukcja struktury 3D neuronu (tzw. mesh) wraz z fragmentem obrazu z mikroskopu elektronowego
Metoda segmentacji struktur 3D neuronów została zaproponowana w pracy Michała Januszewskiego i in. “High-precision automated reconstruction of neurons with flood-filling networks”. (Nature methods, 2018)
W ostatnim etapie konstrukcji mapy wszystkie rozpoznane obiekty warto opisać lub innymi słowy zaanotować, tzn. nanieść poszczególne nazwy ulic, parków, rzek. W neuronauce anotacje zapewniają niezbędny kontekst do interpretacji wzorców połączeń neuronalnych. Znajomość typu komórki i neuroprzekaźnika neuronu pomaga badaczom zrozumieć jego rolę w obwodach neuronowych i funkcjonowaniu mózgu. Ogromna większość anotacji, obejmująca około 100 tysięcy neuronów, była wykonywana manualnie przez małą grupę ekspertów. Do opisu pozostałej części neuronów wykorzystano właściwości symetrii oraz modularności mózgu – struktury analogiczne do zaanotowanych wcześniej łatwiej było opisać na podstawie dotychczasowej pracy. Przy wykrywaniu tych podobieństw w budowie używano algorytmów dopasowywania grafów, wyszukujących takie przyporządkowanie ich wierzchołków, które optymalizuje zgodność krawędzi między nimi. W tym przypadku wierzchołki reprezentowały neurony z dwóch obszarów mózgu, a krawędzie określane były na podstawie liczby połączeń synaptycznych. Tak sformułowane zadanie okazuje się analogiczne do jednego z podstawowych problemów optymalizacji kombinatorycznej: problemu przypisania kwadratowego. Mimo że jest to problem NP-trudny, istnieją jednak efektywne rozwiązania przybliżone dla obu zagadnień, których zastosowanie ułatwiło pracę ekspertom.
W rezultacie, poprzez kombinację narzędzi biologicznych, informatycznych oraz
matematycznych, z sukcesem ukończono projekt FlyWire, o którym pisano w Nature,
czyli największą i najdokładniejszą do tej pory mapę mózgu. Skrupulatnie
zaanotowano 139 255 neuronów organizmu żeńskiego muszki owocówki,
tworzących razem ponad 50 milionów połączeń synaptycznych. Zestaw danych
FlyWire zawiera dwa kluczowe zasoby: morfologie wszystkich neuronów i wykres
sieciowy pokazujący, w jaki sposób łączą się one ze sobą. Co najważniejsze,
projekt jest otwarto-źródłowy, tzn. każdy może zobaczyć rezultat pracy naukowców
na stronie flywire.ai.
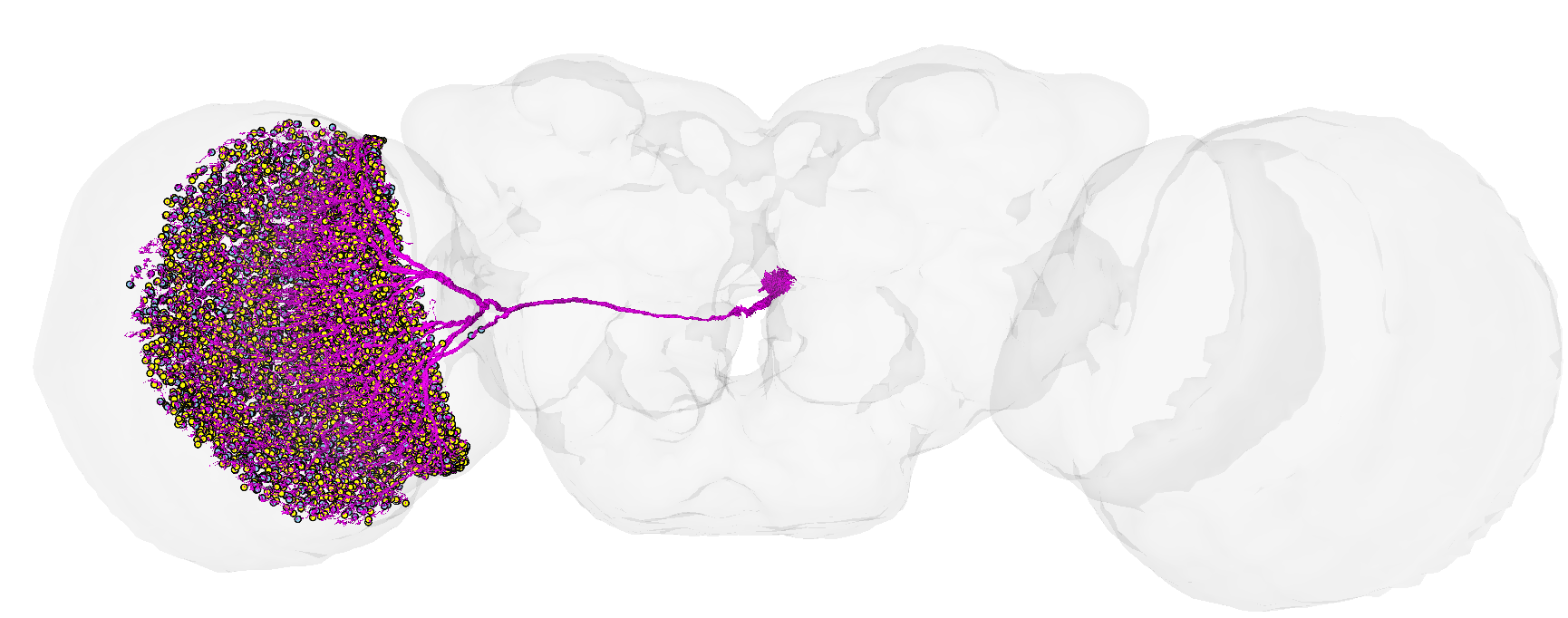
Obrys mózgu muszki owocówki z zaznaczoną rekonstrukcją neuronu wewnętrznego płata wzrokowego. Zrzut ekranu z narzędziaCodexdostępnego naflywire.ai
Mapa ta przyda się w zrozumieniu funkcji struktur mózgowych u muszki owocówki, z których część ma odpowiedniki w mózgu ludzkim, a wypracowane techniki obrazowania i anotacji pomogą przy tworzeniu map mózgu innych, bardziej rozwiniętych organizmów. Ostatecznym celem jest zbudowanie mapy ludzkiego mózgu, do której przechowania (przy precyzji takiej jak FlyWire), jak się szacuje, potrzeba będzie zettabajtów (przedrostek zetta oznacza \(10^{21}\)) pamięci!
Aby film nagrany w standardzie 4K zajmował jeden zettabajt, musiałby trwać około 10 milionów lat.